一組來自 Alphabet ‘ sDeepmind 的計算生物學家利用人工智能從蛋白質的氨基酸序列中預測蛋白質的結構,解開了一個困擾科學家數十年的謎團。
甚至不到一年后,一項新的研究提供了一個更強大的模型,能夠在一臺游戲電腦上在短短 10 分鐘內計算出蛋白質結構。
The research 來自華盛頓大學( UW )的科學家們承諾加快藥物研發,這可以解開治療癌癥等疾病的方法。
存在于身體的每一個細胞中,蛋白質在許多過程中發揮作用,如凝血、激素調節、免疫系統反應、視力以及細胞和組織修復。由長鏈氨基酸相互作用形成折疊的三維結構組成,蛋白質的形狀決定其功能。
未折疊或錯誤折疊的蛋白質也被認為會導致退化性疾病,包括囊性纖維化、阿爾茨海默病、帕金森病和亨廷頓病。了解和預測一種蛋白質結構是如何形成的,可以幫助科學家為許多此類疾病設計有效的干預措施。
華盛頓大學的研究人員通過創建一個同時考慮蛋白質序列模式、氨基酸相互作用和可能的三維結構的三軌神經網絡,開發了 RoseTTAFold 模型。
為了訓練模型,研究小組使用不連續的蛋白質片段,有 260 個獨特的氨基酸元素。具有the cuDNN – 加速 PyTorch 深度學習框架,以及 NVIDIA Geforce 2080 GPU ,這些信息在深度學習模型中來回流動。然后這個網絡就可以推斷出蛋白質的化學成分及其折疊結構。
“ RoseTTAFold 的端到端版本需要在 RTX 2080 GPU 上大約 10 分鐘來生成少于 400 個殘基的蛋白質的骨架坐標。研究人員在研究報告中寫道:“ pyRosetta 版本需要 5 分鐘對單個 NVIDIA RTX 2080 GPU 進行網絡計算,用 15 個 CPU 核生成所有原子結構需要 1 小時。”。
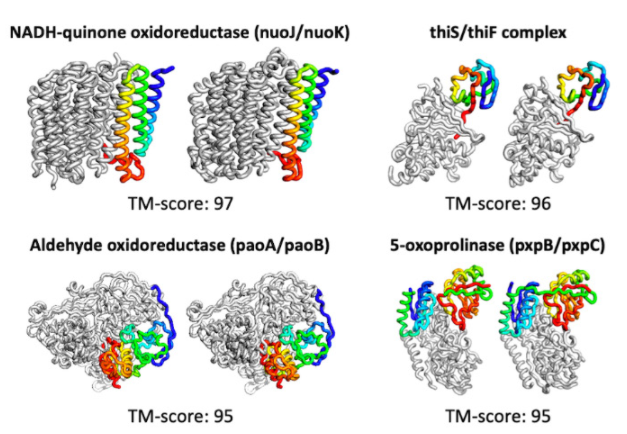
預測蛋白質結構及其基本真值。信貸: UW / Baek 等人
這個工具不僅可以快速預測蛋白質,而且可以在有限的輸入下進行預測。它也有能力超越簡單的結構進行計算,預測由幾個結合在一起的蛋白質組成的復合物。更復雜的模型在 24G 上計算大約 30 分鐘 NVIDIA Titan RTX 。
任何有興趣提交蛋白質序列的人都可以使用公共服務器。這個 源代碼 科學界也可以免費獲得。
“就在上個月,超過 4500 種蛋白質被提交到我們新的網絡服務器上,我們通過 GitHub 網站提供了 RoseTTAFold 代碼。我們希望這個新工具將繼續有益于整個研究社區,”華盛頓大學蛋白質研究所的博士后學者 Minkyung Baek 說。
關于作者
Michelle Horton 是 NVIDIA 的高級開發人員通信經理,擁有通信經理和科學作家的背景。她在 NVIDIA 為開發者博客撰文,重點介紹了開發者使用 NVIDIA 技術的多種方式。
審核編輯:郭婷
-
NVIDIA
+關注
關注
14文章
4940瀏覽量
102815 -
人工智能
+關注
關注
1791文章
46859瀏覽量
237567
發布評論請先 登錄
相關推薦
嵌入式和人工智能究竟是什么關系?
《AI for Science:人工智能驅動科學創新》第6章人AI與能源科學讀后感
AI for Science:人工智能驅動科學創新》第4章-AI與生命科學讀后感
《AI for Science:人工智能驅動科學創新》第一章人工智能驅動的科學創新學習心得
差示掃描量熱儀測試蛋白質的應用案例

risc-v在人工智能圖像處理應用前景分析
創客中國AIGC專題賽冠軍天鶩科技:AI蛋白質設計引領者

EvolutionaryScale推出基于NVIDIA GPU模型的新型蛋白質研究方案
報名開啟!深圳(國際)通用人工智能大會將啟幕,國內外大咖齊聚話AI
FPGA在人工智能中的應用有哪些?
洪亮團隊在生信期刊JCIM發布最新成果,蛋白質工程邁入通用人工智能時代
對新輔助TCHP治療響應的HER2+乳腺癌空間蛋白質組特征





 利用人工智能技術預測蛋白質的結構
利用人工智能技術預測蛋白質的結構










評論