作為一名對醫療應用人工智能感到興奮的本科生,我很高興能加入 NVIDIA Clara 部署團隊進行實習。這是一個完美的組合:有機會在一家領先的技術公司工作,加速和采用人工智能,同時為未來(和現在)的團隊建設做出貢獻用于醫療保健的人工智能部署。接下來的幾個月充滿了從才華橫溢但謙遜的同事那里學到的東西,學習了 CUDA 編程等新技能,以及關注組織病理學數據帶來的獨特技術挑戰的機會。
什么是 Clara Deploy?
Clara Deploy SDK 是一個基于容器的云本機開發和部署框架,用于智能醫院中的多 AI 和多域工作流。它使您能夠定義由多個階段組成的基于容器的管道,每個階段由操作員定義。管道由多個運算符組成,是從數據源到數據接收器的有向無環圖( DAG )。每個操作符都是管道的一個步驟,比如加載輸入、預處理、人工智能推理等等。
在我探索建立 NVIDIA Clara Deploy 平臺和運行 AI 推理管道的過程中,我獲得了部署 AI 工作流挑戰的第一手經驗,特別是在標準化工作流和擴展執行方面。在運行數字病理管道的過程中,我意識到了 I / O 和預處理步驟的性能瓶頸,這些步驟通常不會 GPU 加速。這影響了我在實習期間專注于加速數字病理學預處理過濾器的選擇。
什么是 cuCIM ?
cuCIM 是一個 RAPIDS 庫,用于加速 n 維圖像處理和圖像 I / O ,重點是醫學成像應用。 cuCIM 由 I / O 、文件系統和操作模塊組成。 cuCIM 中的操作可以使用插件架構進行擴展。 cuCIM 在醫學圖像處理應用方面處于領先地位,我很高興在 NVIDIA 工作期間接觸并參與其中。
項目動機
組織病理學分析數字化的一個重大挑戰是病理學圖像中觀察到的染色變化。這些圖像可能由多種因素引起 染色差異大 ,包括染色供應商、儲存條件、染色協議、數字掃描儀等。
鑒于因素的范圍,在圖像采集期間控制染色變化是不切實際的。取而代之的是,一個稱為染色標準化的圖像預處理步驟通常用于算法標準化圖像染色。染色規格化過濾器接受源圖像和目標圖像作為輸入。將源圖像進行染色歸一化,目標圖像包含理想染色,并將其轉移到源圖像。最終,標準化的源圖像作為輸出返回。
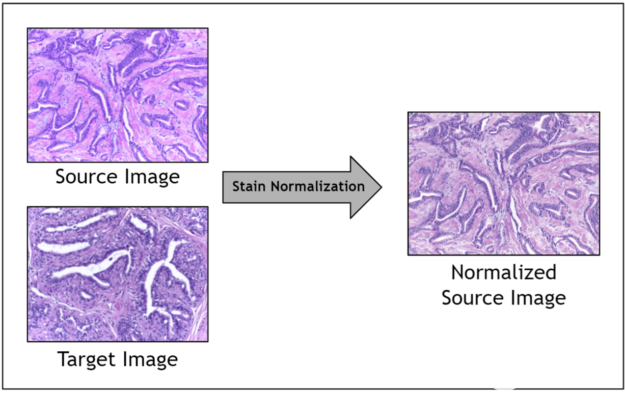
圖 1 。染色標準化過濾器。來自 StainTools 。
先前的工作表明,在數字病理 AI 管道中,作為預處理步驟的染色歸一化可以縮短訓練時間,提高準確性,并使來自不同來源的數據能夠一起使用。由于染色病理圖像的稀缺性,您在相對較小的數據范圍內操作,因此染色標準化使您能夠優化在噪聲染色變化中獲得的信號。
然而,之前的染色標準化實施相對緩慢,因為它們沒有 GPU 加速。有機會實現 GPU 加速染色歸一化算法,并為數字病理 AI 管道實現快速有效的預處理。
加速數字化病理學染色標準化
染色標準化方法分為三大類:
全局顏色歸一化
色斑反褶積后的顏色歸一化
基于深度網絡的顏色傳遞
有關更多信息,請參閱 染色顏色自適應歸一化( SCAN )算法:數字病理學中組織染色的分離和標準化 。
我選擇專注于基于染色反褶積的方法,因為之前的文獻顯示,與基于深度網絡的方法相比,與全局顏色歸一化相比,其性能更高,并且在維持生物結構完整性方面具有更好的理論保證。
基于染色反褶積的方法假設每個圖像都具有染色矩陣的特征,該矩陣包含 H & E 染色圖像中兩種染色(蘇木精和曙紅)的紅、綠、藍( RGB )值。
使用 比爾 – 蘭伯特定律 將 RGB 圖像轉換為光密度圖像。然后,光密度圖像可以與該圖像的像素濃度矩陣和染色矩陣的乘積相關。像素濃度矩陣表示每個像素的每個染色的濃度。如果使用 馬森科法 估算染色矩陣,則可獲得濃度矩陣。
最后,對于染色歸一化,源圖像的染色矩陣被替換為 目標圖像的染色矩陣 。這用于將染色輪廓從目標圖像傳輸到源圖像。因為源圖像的濃度矩陣不變,所以生物結構的形態保持不變。用于估計染色矩陣的 Macenko 方法是一種使用 奇異值分解 的無監督方法。
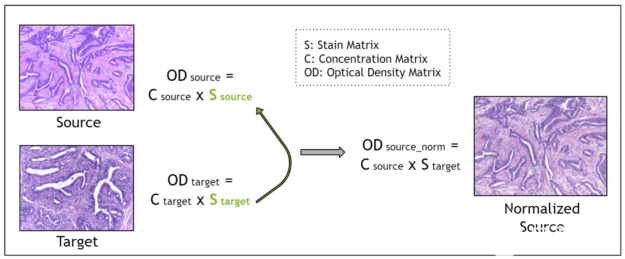
圖 2 。基于應變反褶積的應變歸一化
在修改了 NumPy 中的現有版本后,我為 CuPy 中的 Macenko 染色標準化方法設計并實現了一個過濾器。接下來,我比較了兩者的性能。
圖 3 顯示了使用 NVIDIA DGX-1 對不同圖像大小的 NumPy 和 CuPy 實現的著色歸一化的相對性能。 CuPy 實現的性能根據相對于 NumPy 實現的加速因子繪制。
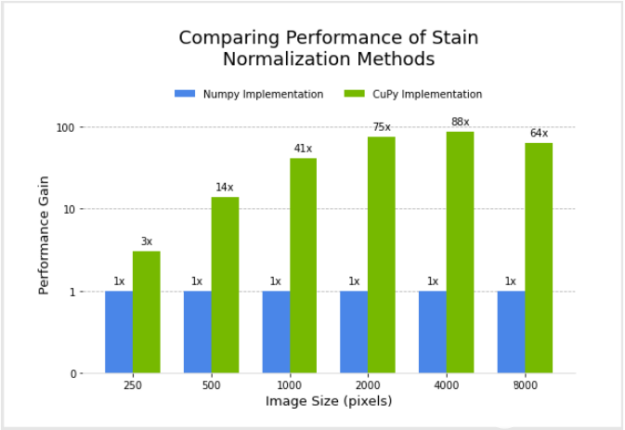
圖 3 。 NumPy 與 CuPy 染色標準化實現的性能比較
考慮到使 GPU 加速染色標準化能夠用作數字病理學管道的預處理步驟的目標,我開始將該過濾器作為轉換(基于陣列和基于字典)集成到 MONAI 。 MONAI 是一個基于 PyTorch 的開源框架,用于醫學成像的深度學習。完全集成后,可以將染色歸一化變換添加到 Clara Train 或 MONAI 中的病理管道中。
彩色轉換濾波器的加速
接下來,我在 CUDA C ++中實現了顏色轉換 RGB2HED 函數,這是一個常用的函數,在 scikit-image 和 Cuim-Python 層中可用。從 RGB 到 HED 的顏色空間轉換與色斑歸一化密切相關,因為該函數涉及獲得色斑濃度值,假設色斑向量是一個恒定的、預先計算的近似值。這忽略了不同圖像染色之間的差異。這個功能通過 C ++的操作符插件機制集成到 CuCIM 中。
我比較了純 C ++實現和 CUDA C ++實現的性能。圖 4 顯示了使用 NVIDIA GV100 GPU 和 Intel ( R ) Core ( TM ) i7-7800X CPU 的兩個版本在不同圖像大小下的相對性能。 CUDA C ++實現的性能以相對于純 C ++實現的加速因子的形式繪制。
需要注意的是,性能提升并沒有考慮到與 GPU 之間的任何數據傳輸。我之所以這樣做,是因為我正在考慮一種常見的場景,即在圖像處理工作流中的幾個后續操作中,數據傳輸通過留在 GPU 上而最小化,并且只在最后發生向主機的傳輸。
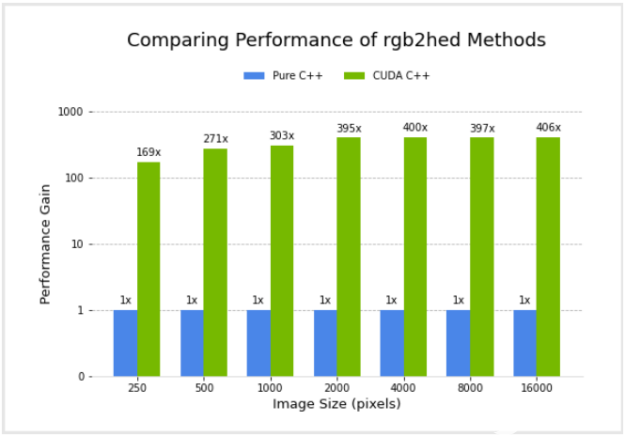
圖 4 :純 C ++與 CUDA C ++實現的性能比較 rgb2hed 顏色轉換函數
結論
總之,我的實習項目的重點是加速數字病理學的顏色轉換過濾器。具體來說,我致力于設計和實現 Macenko 染色歸一化方法,使用 CuPy 進行 GPU – 加速。我開始將其集成到 MONAI 中,作為一種轉換,以便將來用作數字病理管道的預處理步驟。接下來,我在 CUDA C ++中實現了顏色轉換 RGB2HED 函數,通過基于 C ++的操作符插件機制將其集成到 CuCIM 中。
Macenko 染色規范化和 CUDA C ++實現的 RGB2HED 函數都表現出顯著的性能增益,與 NumPy 版本和純 C ++版本相比,它們都表現出顯著的性能增益。使用基于 NumPy 的過濾器,在 250 個圖像數據集和 4000 × 4000 像素圖像大小的情況下,訓練 500 個時代以上的管道的染色歸一化預處理時間大約為 13 天。 CuPy 基過濾器的過濾時間減少到 3.5 小時。
最終,加速數字病理學的前處理和后處理過濾器可以提高數字病理學深度學習管道的性能,加快數字病理學的采用,并使人工智能能夠徹底改變病理學。
關于作者
Neha Srivathsa 是 NVIDIA 的 Clara Deploy 應用程序團隊的實習生。在實習期間,她一直致力于 GPU ——數字病理管道預處理過濾器的加速。 Neha 是斯坦福大學的一名本科生,研究計算機科學,重點是人工智能和生物信息學。
審核編輯:郭婷
-
NVIDIA
+關注
關注
14文章
4940瀏覽量
102817 -
計算機
+關注
關注
19文章
7423瀏覽量
87719 -
C++
+關注
關注
22文章
2104瀏覽量
73497
發布評論請先 登錄
相關推薦
一文理解布隆過濾器和布谷鳥過濾器

CH32FV系列CAN設備過濾器配置

PLC水處理過濾器運維管理系統解決方案
PLC工業過濾器數據采集物聯網解決方案

康謀分享 | ADTF過濾器全面解析:構建、配置與數據處理應用

信號分析和過濾器的作用
康謀技術| 揭秘汽車功能的核心——深度解讀ADTF中的過濾器圖
AN-B-099:DA14535 減少凈排放過濾器應用說明





 使用NVIDIA Clara Deploy加速數字病理學預處理過濾器的選擇
使用NVIDIA Clara Deploy加速數字病理學預處理過濾器的選擇










評論