自單細胞測序技術橫空出世以來,由于其可以將測序縮小到單個細胞的單位內進行,對于更加全面精確地了解生物基因表達情況具有重要意義。但是,由于單細胞測序本身的技術特點,盡管我們可以得到單個細胞的表達信息,但是細胞在原本組織內的空間位置信息是完全丟失的,這些信息在理解細胞微環境或細胞間互作時同樣關鍵。
為了解決這個問題,空間轉錄組技術出現了,這項技術可以在組織原位上以接近單個細胞大小的尺度來進行轉錄組測序,但因為技術限制以及生物組織本身的復雜性,空間轉錄組還不能精確提供單個細胞尺度上的轉錄組信息。
單細胞測序技術具有單個細胞的精度,空間轉錄組提供了組織原位的信息,因此將兩者結合起來就能近似得到完整的信息。目前已經有許多分析工具試圖將空間信息與單細胞信息整合起來,并且取得了一定的進展。本文將介紹的CellTrek就是其中之一。
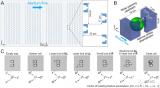
目前常用的整合算法有許多,例如去卷積,通過推斷spot內的細胞組成來將兩者整合在一起。而CellTrek則另辟蹊徑,通過直接推斷單細胞的空間坐標來實現信息的補全。Cell Trek的計算原理可見下圖。簡單來說,該方法首先將單細胞數據與空間轉錄組數據整合,共嵌入到(coembed)共同的隱空間內,然后從中抽出空間轉錄組的數據作為訓練數據集,使用隨機森林模型(Random Forests,RF)從spot的基因表達情況預測其空間坐標。這里所依據的基本思路就是,細胞在組織中的分布并不是隨機混亂的,同一類細胞大多都是集中于同一片區域。
空間轉錄組的數據還會同時進行非線性插值增強其空間分辨率。然后,將整個共嵌入矩陣輸入訓練好的模型,生成一個衡量單細胞與空間spot之間表達相似性的RF距離矩陣,基于這個矩陣使用相互最近鄰域算法(mutual nearest neighbors,MNN)生成一個spot-細胞稀疏圖,最后根據細胞在稀疏圖上的相鄰spot推斷出細胞的空間坐標。因為相同類型的細胞擁有較為相似的基因表達,所以基于空間轉錄組數據訓練的模型可以用來預測細胞的位置坐標。
01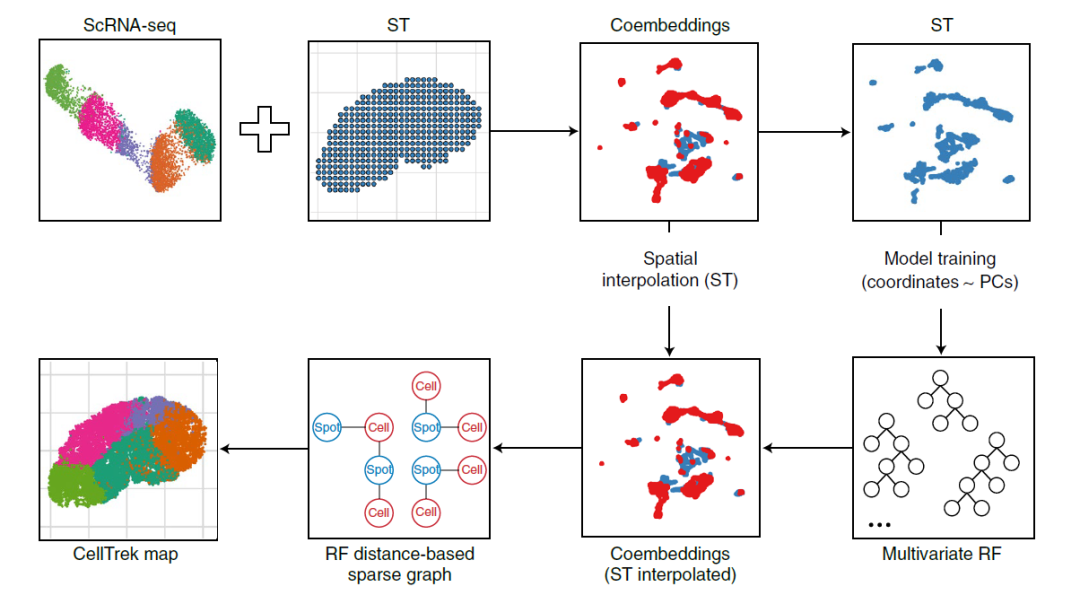
這一算法的優勢在于將細胞直接定位到組織切片照片上,而不是像去卷積類方法那樣計算每個spot內的細胞類型比例。直接定位帶來的另一個好處就是在接下來的數據分析中可操作性大大提高了,像去卷積類方法得到的細胞比例矩陣我們很難再對其進行什么操作,但是我們可以對CellTrek 整合后的結果進行細胞共定位分析、基因空間權重共表達分析等等,因為他本質仍然是單細胞數據,只是附帶上了空間坐標。
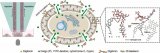
以小鼠大腦皮層的空轉數據與單細胞數據為例,我們最后得到的結果會是這樣的。可以看到不同類型的細胞在空間上呈現出明顯的層狀分布,這符合大腦皮層的結構特征,并且不同的細胞類型出現在了它們應該出現的皮層位置。 02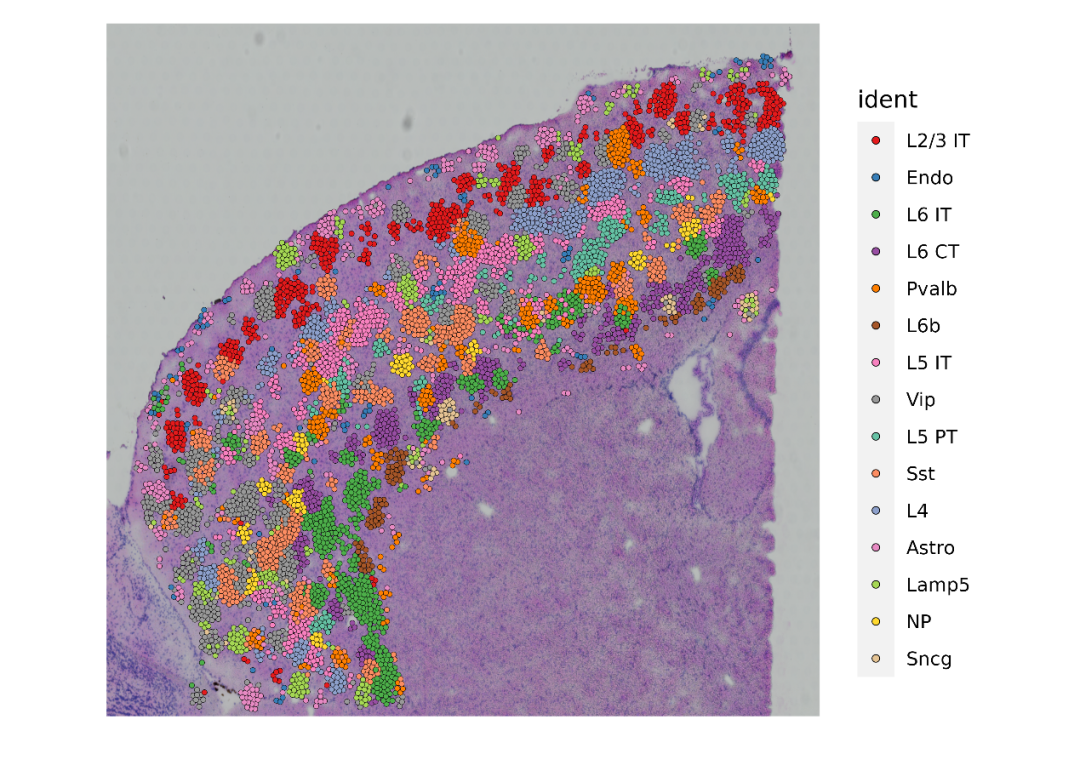
內測數據結果
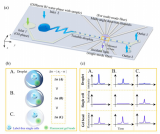
CellTrek的缺點在于,第一,對計算資源的需求比較大,當單細胞數據量較大時會花費比較長的時間;第二,對于空轉數據與單細胞數據的匹配度要求比較高,也就是說空轉數據與單細胞數據最好取自同一塊組織區域,不要出現無法匹配的空間區域或細胞類型,否則最終結果會非常混亂,如下圖。
03
內測數據結果
總而言之,CellTrek提供了一條不同于當下其他單細胞-空轉整合分析方法的道路,結果更加接近生物組織中的實際情況,并為整合后的下游分析提供了便利。
審核編輯:劉清
-
RF信號
+關注
關注
1文章
41瀏覽量
14651 -
卷積網絡
+關注
關注
0文章
42瀏覽量
2158
原文標題:單細胞空間轉錄組聯合分析硬核出擊--CellTrek
文章出處:【微信號:SBCNECB,微信公眾號:上海生物芯片】歡迎添加關注!文章轉載請注明出處。
發布評論請先 登錄
相關推薦
寬帶功率放大器基于微流控技術的細胞分選的應用
基于啟揚 i.MX8M Mini核心板的全自動細胞分析儀應用解決方案

基于雙極性電極陣列的微流控芯片,可實現細胞可控、非接觸三維旋轉

gis中常用的空間分析方法
arcgis空間分析方法有哪些
采用LIBS成像技術研究納米顆粒在單細胞內的亞細胞分布

復旦大學郝潔/鄒欣團隊Cell子刊發表單細胞分析新技術用于腫瘤精準免疫治療
東南大學在釀酒酵母單細胞復制衰老壽命圖像分析算法方面取得進展
百篇文獻匯總精華!單細胞高級分析(四)





 CellTrek單細胞空間轉錄組聯合分析
CellTrek單細胞空間轉錄組聯合分析











評論