空間轉錄組學技術是揭示組織精細解剖位置的組學工具。基于高通量測序的空間轉錄組學技術具有檢測結果無偏移、使用門檻低等優勢。目前,科學家開發了多種技術構建條形碼RNA捕獲陣列,然而難以實現成本、探針編碼復雜度、基因捕獲靈敏度和空間分辨率之間的平衡關系。
近日,吉林大學張俊虎、梁重陽團隊在預印本bioRxiv上發表了題為“Well-ST-seq: cost-effective spatial transcriptomics at cellular level and high RNA capture efficiency”的文章,報道了一種低成本、細胞分辨率水平、高RNA捕獲效率的空間轉錄組測序(ST-seq)方法。
該技術將帶有高密度活性基團的微珠排列于微井陣列,再采用微流控技術對微珠陣列進行編碼,生成高密度的RNA捕獲探針,其靈敏度可達3896 UMIs/10微米。該探針陣列制備全程未用到大型精密儀器,具有小型化、低成本優勢,構建成本約為1.21美元/mm2。
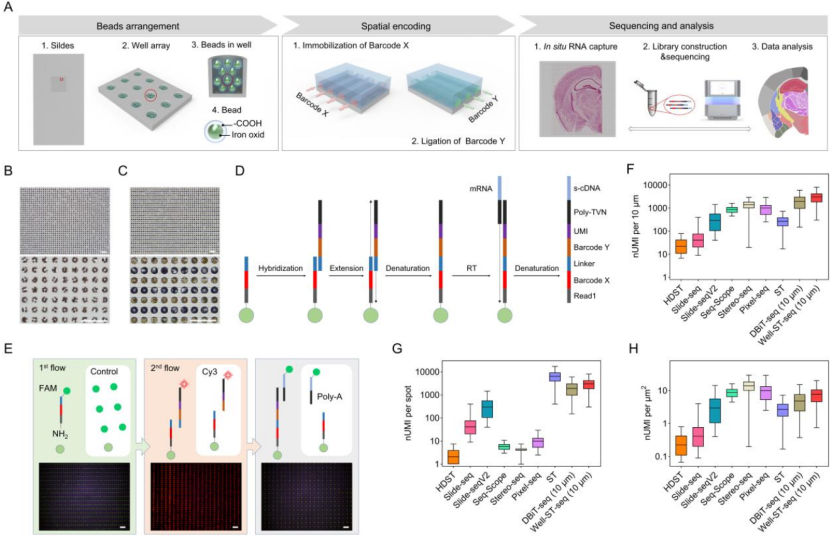
Well-ST-seq技術概述
此外,該技術空間分辨率由微孔道及微井陣列尺寸控制,可實現10微米分辨率的空間轉錄組測序。
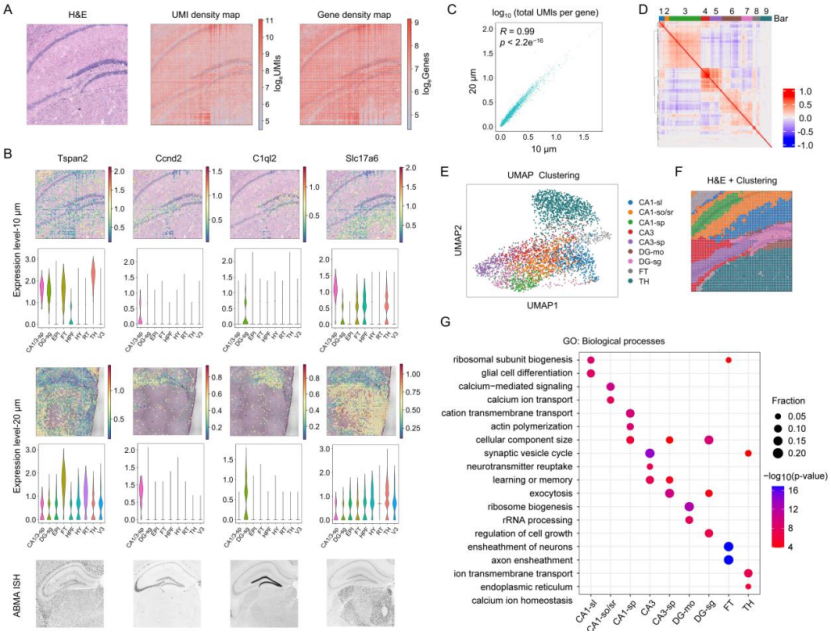
通過Well-ST-seq-10 μm繪制小鼠海馬的高空間分辨率圖譜
為了驗證Well-ST-seq在高分辨率空間轉錄組測序方面的靈活性,研究團隊制作了10 μm分辨率的微通道和微孔陣列,并選擇小鼠大腦的海馬區域進行組織切片。Well-ST-10 μm和Well-ST-20 μm數據中標記基因的空間表達圖譜顯示出了高度的一致性。Well-ST-10 μm檢測到的標記基因表達與Allen小鼠腦圖譜(AMBA)的ISH數據一致,進一步驗證了Well-ST-seq的準確性。
而與Well-ST-seq-20 μm數據相比,Well-ST-seq-10 μm生成的空間地圖顯示出更詳細的解剖形態,與組織切片的HE染色圖像一致;與基于ABMA注釋結果的解剖學定義的腦區很好地對應,并且集群的標記基因表現出差異表達模式。結果表明,Well-ST-seq能夠進行高分辨率的空間轉錄組學分析。
總而言之,該技術為空間單細胞轉錄組測序提供了低成本的解決方案,且實現了高靈敏度、分辨率、可伸縮性以及與現有數據庫的兼容性,是一種高效、經濟、便利的原位捕獲和分析方法,為空間轉錄組學研究開辟了新的可能性。
審核編輯:劉清
-
微流控芯片
+關注
關注
13文章
249瀏覽量
18747 -
RNA
+關注
關注
0文章
46瀏覽量
9682 -
微流控系統
+關注
關注
1文章
60瀏覽量
1840
原文標題:基于微流控的低成本空間轉錄組測序方法:細胞分辨率水平、高RNA捕獲效率
文章出處:【微信號:Micro-Fluidics,微信公眾號:微流控】歡迎添加關注!文章轉載請注明出處。
發布評論請先 登錄
相關推薦
微流控芯片在生物學有何應用?微流控芯片微液滴、檢測技術介紹
專為京東方科技集團股份有限公司研發的測序芯片、制備方法和測序裝置





 基于微流控的低成本空間轉錄組測序方法
基于微流控的低成本空間轉錄組測序方法












評論