摘要:研究人員首次將深度學習與貝葉斯假設檢驗結合,利用深度學習強化RNA可變剪接分析的準確性。
在生命科研領域,常有人說深度學習的基因組學應用好比是“一個盲人在一間黑暗的房子里尋找一頂并不存在的黑色帽子”。言下之意,是遺憾深度學習的基因組學應用并沒有給人們帶來太多驚喜。不過,近日賓夕法尼亞大學和費城兒童醫院教授邢毅團隊的一項研究,找到了這樣一頂“黑帽子”。
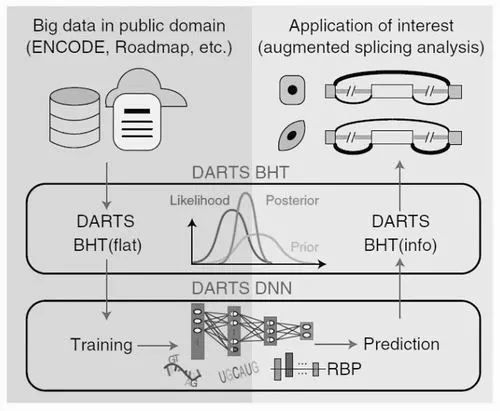
這項發表在《自然—方法》上的論文成果,提出了一種新的計算框架——DARTS(“利用深度學習強化對RNA-seq的可變剪接分析”英文的首字母縮寫)。該計算框架首次將深度學習與貝葉斯假設檢驗結合,用于RNA可變剪接分析。這種結合使得它即使對于測序深度不那么高的樣品,也能有效提高RNA-seq定量差異剪接的準確度。
清華大學生命科學學院教授張強鋒點評道:“DARTS綜合了深度學習和貝葉斯假設檢驗統計模型的優點,為那些低測序深度的數據提供了更好的做可變剪接分析的手段,拓展了傳統RNA-seq可變剪接分析的敏感度和準確度。”
計算基因組學中
一個廣受關注的問題
邢毅等人在上述論文中指出,目前,RNA-seq技術是研究RNA剪接最常用的實驗手段。然而,RNA-seq技術雖然能較好地定量基因表達的結果,但對于差異剪接分析來說,它依賴于更高的測序深度。而且即便如此,現有的計算方法還不能較準確地定量低表達基因的剪接變化。因此,為了提高剪接定量的準確性,急需引入新的計算分析方法。
“可變剪接現象從20世紀70年代被發現后,其基本的科學問題聚焦為可變剪接位點發現、差異分析、調控元件和網絡的發現和構建。RNA-seq 技術的發明,使得系統、定量的可變剪接差異分析成為可能。”張強鋒介紹說,大量測序數據的可變剪接差異分析需要優秀的統計模型和計算工具,因此一直是一個需要高度技巧的生物信息學研究課題。
據張強鋒介紹,邢毅研究組在針對大量測序數據的可變剪接差異分析的計算分析領域深耕多年,已經貢獻了多個有影響力的算法和計算工具。該團隊針對高通量RNA-seq數據開發出的用于差異剪接分析的rMATS等軟件,對于測序較深、質量較好的數據集都能取得不錯的結果,已在全世界范圍內被廣泛下載使用。
然而,由于成本等原因,大量RNA-seq 測序實驗設計的測序深度較淺。對于這些數據集,能利用來做差異分析的可變剪接事件非常有限。
美國卡耐基梅隆大學計算機學院教授馬堅也表示,在基因組學中,確實有很多類似的問題——如何在現有數據上對特定的基因組標注(譬如染色質結構、轉錄因子結合)訓練一個機器學習模型并在全新的細胞系中有效預測,已經成為一個計算基因組學中廣泛關注的問題。“DARTS嶄新的整體設計理念值得很多其他類似的問題借鑒。”
DARTS計算框架
給出問題答案
據邢毅研究組這篇發表在《自然—方法》上的論文介紹,DARTS由兩部分構成:深度神經網絡模塊(DNN)和貝葉斯推斷模塊(BHT)。其中,DNN基于順式序列特征和樣品特異的RNA結合蛋白表達水平特征來預測差異剪接的結果;而BHT則通過整合實驗樣品測序數據本身和基于深度神經網絡的先驗概率來推斷差異剪接的結果。
研究者在論文中強調稱,與其他計算方法不同的是,在DARTS計算框架下,DNN不僅通過順式序列特征來預測可變剪接的結果,而且還將樣品中RNA結合蛋白的表達水平整合進了RNA可變剪接結果的預測中,增加了預測參數的維度。
DARTS的邏輯是,通過DNN對ENCODE和Roadmap數據庫中大量RNA-seq結果的深度學習,能夠獲得高精度的預測值作為BHT中的貝葉斯先驗概率,進而結合具體實驗中RNA-seq的結果,來獲得更為準確的差異剪接推斷。
在研究實踐中,邢毅研究組發現,在低通量RNA-seq文庫中,通過使用DNN預測值進行強化分析后,能夠達到比使用傳統方法分析更高的準確度,并且這種提升在越低通量的文庫中越明顯;即使在高通量的RNA-seq文庫中,使用DNN預測仍能發現在低表達基因中的可變剪接變化。而在過去,這些低表達基因的可變剪接變化在傳統分析方法中往往會被忽略。
也就是說,研究結果證明了DARTS不僅提升了基于RNA-seq方法研究可變剪接的準確性,同時也提供了在低表達基因中研究可變剪接的研究手段。
解析DARTS:
海量數據訓練和新樣本特征的綜合
“從計算方法設計的策略和概念角度而言,此工作的最大亮點是充分利用海量公有數據如ENCODE,但模型本身又不完全依賴于這些公有數據。”馬堅點評道,換言之,DARTS的整體思想是用深度神經網絡從現有海量數據中找出通用的有用信息作為先驗,然后用貝葉斯假設檢驗結合來自樣本本身的RNA-seq數據信息,做可變剪接的預測,“這有效綜合了海量數據的訓練以及新樣本的特殊性”。
馬堅解釋說,從模型本身的技術角度而言,DARTS有效利用了深度神經網絡對異質數據特征的整合,并且整個計算方法的評測和方法都比較“明智而審慎”。他舉例說,比如DARTS的深度神經網絡部分結合了剪接位置附近的序列信息、進化信息、可變剪接產生的RNA二級結構信息等;同時DARTS還巧妙地利用深度神經網絡預測的結果來作為貝葉斯假設檢驗中的先驗數據,結合樣本本身的RNA-seq序列信息實現了更可靠的可變剪接預測。
馬堅將基因組學形容為一個“存在太多未知和容易迷失的領域”,因此他認為,有效深度學習的使用需要有強大的領域知識作為支撐。而DARTS工作恰恰體現了邢毅實驗室多年以來對可變剪接機理的研究和計算方法創新的積累。“由深入的領域知識和經驗作為指導,是一個有效利用不同計算模型和深度學習方法的優勢實現基因組學新發現的經典工作。”
張強鋒也直指“巧妙利用公開的RNA-seq大數據樣本、使用深度神經網絡學習得到了外顯子差異剪接的貝葉斯假設檢驗統計模型的準確先驗概率分布”是DARTS在方法上最大的特色。同時他也表示,該計算框架使用深度神經網絡通過順式序列和反式因子RBP表達豐度進行差異剪接預測的思路也值得借鑒。
此外,馬堅認為論文中其他對于機器學習方法的評測同樣可圈可點。例如,對常見的正負樣本不均衡的問題對模型訓練和評測可能帶來的偏差有細致的控制。另外,該計算框架對模型中每個模塊的貢獻也做了詳細分析。
“隨著RNA-seq數據的不斷積累,相信DARTS會有廣泛的應用,尤其是在RNA-seq測序深度并不高的實驗情況下。”馬堅說,這個計算工具對進一步理解可變剪接在不同細胞狀態下的調控機理有深遠的意義。
-
數據
+關注
關注
8文章
6892瀏覽量
88828 -
深度學習
+關注
關注
73文章
5492瀏覽量
120977
原文標題:科學家找到深度學習基因組學應用的一頂“黑帽子”
文章出處:【微信號:AItists,微信公眾號:人工智能學家】歡迎添加關注!文章轉載請注明出處。
發布評論請先 登錄
相關推薦
海量數據處理需要多少RAM內存
什么是協議分析儀和訓練器
Llama 3 模型訓練技巧
【《大語言模型應用指南》閱讀體驗】+ 基礎知識學習
海上電磁干擾訓練系統
BP神經網絡最少要多少份樣本
pytorch如何訓練自己的數據
神經網絡如何用無監督算法訓練
機器學習中的數據預處理與特征工程
人臉識別模型訓練是什么意思
特征工程與數據預處理全解析:基礎技術和代碼示例





 解析DARTS:海量數據訓練和新樣本特征的綜合
解析DARTS:海量數據訓練和新樣本特征的綜合










評論